主页:https://web.rniapps.net/metagenonets/

副标题:微生物相关网络的推理和整合见解
特色:一站式平台,可从丰富数据中推断,分析,比较和可视化微生物关联网络
主页上方有示例、提交和教程三个按钮
简介
什么是MetagenoNets?
MetagenoNets的名称源于微生物网络生物学中的两个非常普遍的需求:
1)根据环境(例如疾病状态)的丰富程度推断其“微生物关联/网络”
2)根据全面的“元数据”,对所有小组级别(例如健康、对照、感病)的网络进行分别分析;推断与“连续元数据类别”和所有组间关联的相关性(给定相同环境的二级组间丰度概况)。
当前的局限性和挑战是什么?
冗长的工作流程,需要进行丰富的配置文件、推断见解和可视化:
典型的工作流程涉及(a)数据过滤以去除虚假或不相关的特征(b)从多种数据归一化和转换策略中进行选择以考虑样本间偏差,混杂因素,组成等(c)在多种相关推断方法中进行选择导出网络文件(相关矩阵,邻接矩阵,边列表,gmls,jsons)(d)使用图论算法来计算网络特征(例如全局网络属性,局部中心度量)(e)使用可视化包查看网络
元数据引入了额外的复杂性:
与宏基因组学研究相关的全面元数据的可用性为推断和探测微生物关联网络的问题增加了另一层复杂性。对于给定的宏基因组环境,可以有多个级别的元数据组(例如,地理环境可以将国家作为组)。这就需要为每个这样的组分别处理网络。此外,经常还会收集连续的元数据(如BMI,年龄),因此研究人员也有兴趣探索微生物丰度与此类连续数据点的相关性。
组间数据进一步增加了复杂性:
宏基因组学研究通常具有一个或多个“相关”的组学内丰度图。例如,WGS研究不仅可以为研究人员提供微生物的丰度概况,还可以为各种功能单元(如酶,GO,COG,基因等)提供丰度。对相同样品进行的组学研究(如基因表达谱分析)也可以成为紧密相关的组学数据。16S研究的推论功能是与微生物丰度图谱相关的组间图谱的另一个示例。此类二级数据集的可用性通常导致需要找到微生物谱与此类组间单元(如功能,基因等)的相关性。这种相关性的结果通常以“组间整合网络”和“二分网络”的形式可视化。然而为每个元数据类别(和相应的组)实现相同的过程非常繁琐。
MetagenoNets如何应对挑战?
本次介绍MetagenoNets Web服务器是为了简化推断和分析相关驱动的微生物关联网络的过程。MetagenoNets的以下功能有望为微生物组网络分析领域带来巨大的附加价值:
(a)接受所有类型的小型到大型微生物特征表以及多级元数据。提供辅助功能表可为整合分析提供更深入的见解。
(b)提供常用的数据标准化方法(TSS,CSS,四分位数)和转换策略(DeSeq2,TMM,CLR)
(c)通过基于流行和发生的过滤器来减少特征
(d)全面提供主要的最新相关驱动网络推理方法(i)SPARCC(ii)ReBoot / CCREPE(iii)NAMAP(改进的ReBoot),以及基于Pearson和Spearman系数的自举(bootstrap)和经典方法
(e)将元数据智能分类为分类数据和连续数据类型,从而允许最终用户通过集成和二分网络将连续元数据与主要特征集自动关联
(f)三种类型的网络生成选项:(i)对各个单独的网络进行分类或分组(ii)元数据和/或辅助数据集成网络(iii)二分网络,从而能够全面使用丰度数据集以及相关元数据
(g)网络和网络属性的交互式可视化
(h)通过交互的维恩图和交叉点的网络图对分类元数据驱动的网络组进行基于集合的比较
(j)易于使用的用户界面和现代网页设计方法,可在前端实现无缝体验
MetagenoNets方便最终用户同时推断,统计分析和比较相关性驱动的微生物关联网络,并在此过程中以自动化方式生成大量直观、容易解读的可视化输出。
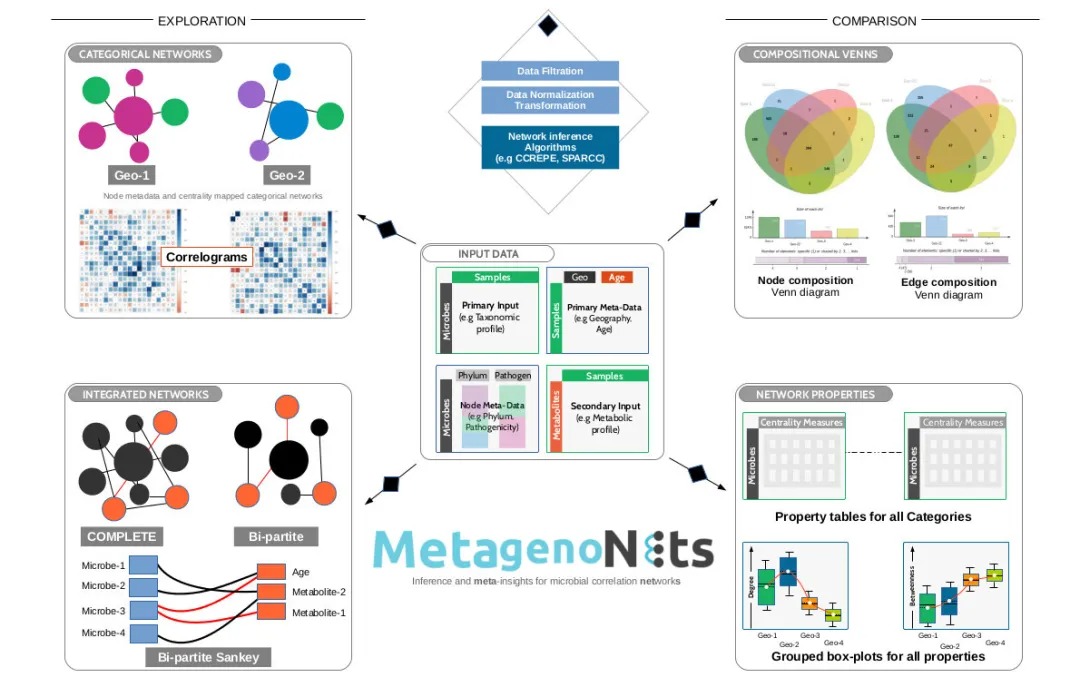
图. 网站的主要结果示例
浏览器兼容性
支持关注主流的浏览器。
浏览器
系统
Firefox v.71 | Linux, Windows, Mac |
Chrome v.79 | Linux, Windows, Mac |
Safari v.12 | Mac |
演示数据和格式介绍
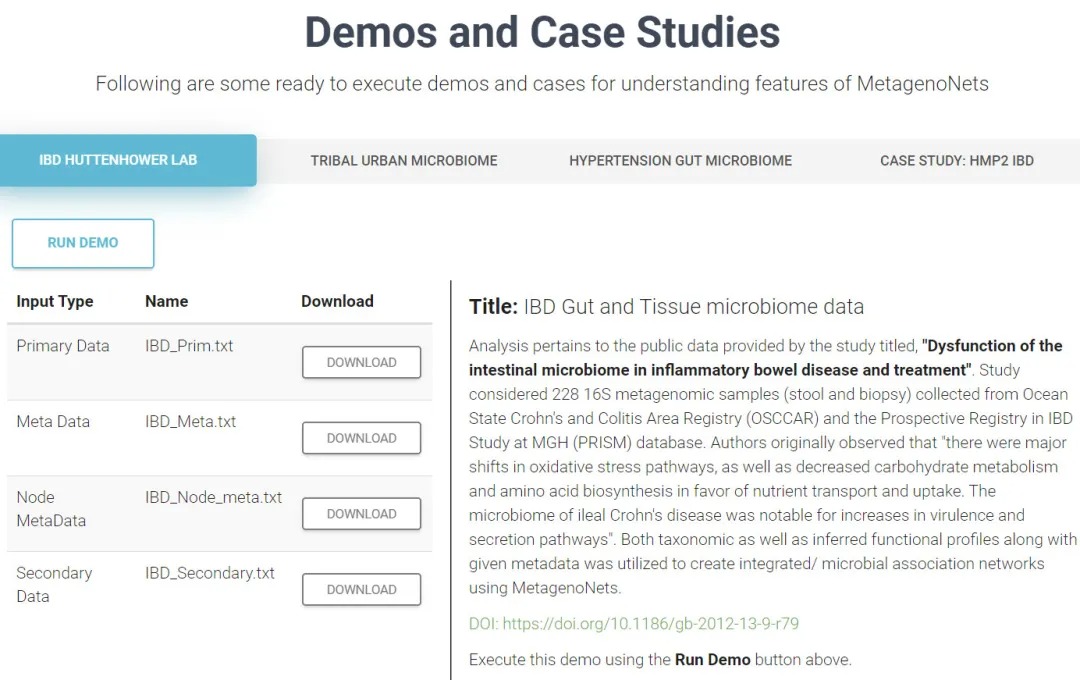
共有4套数据集,分别来自Huttenhower实验室的IBD数据、现代和部落人肠道菌群、高血压肠道微生物组、HMP2的IBD数据。
以第一组IBD为例进行详细介绍和演示。
输入文件:
主要数据(Primary Data):为物种组成特征表,通常为属水平的读长计数(reads count)值;
元数据(Meta Data):元数据,即样本的分组信息和其他属性,如疾病状态、性别、样本类似以及测试的代谢物含量;
节点元数据(Node Metadata): 为属对应的更高级分类,包括门、纲、目、科
次要数据(Secondary Data):样本对应的功能组成表。
点击Next或上方实验名称切换不同数据。点击RUN DEMO按扭运行此数据。
运行测试数据

网络分析的基本步骤:
在左侧选择实验组,并点更新(UPDATE)
等待几秒会显示计算好的结果;
如果要整合连续型数据,或多组学数据,切换至整合网络(Integrated Networks);
选择连续型 或 多组学特征整合二分网络。
分类网络(Categorical networks)
CHOOSE NETWORK TO CREAT 是选择分组,如CrD / Healthy,这里选择 Healthy ,点Update更新网络。
此外面方还有参数可选:
算法Algorithm:默认为Spearman,可选Pearson, SparCC等;
p-value:默认为0.05,边多可进一步减少为 0.01,0.005(不能选更小了);
迭代(Iterations):默认100,0-500次,Sparcc需要5分钟运行;
相关阈值(Corr. cutoff): 默认为严格r-value。可手动设定阈值筛选Spearman, Pearson, CCLasso的相关。
修改参数后,要点击 PROPAGATE TO Modules 来更新网络。
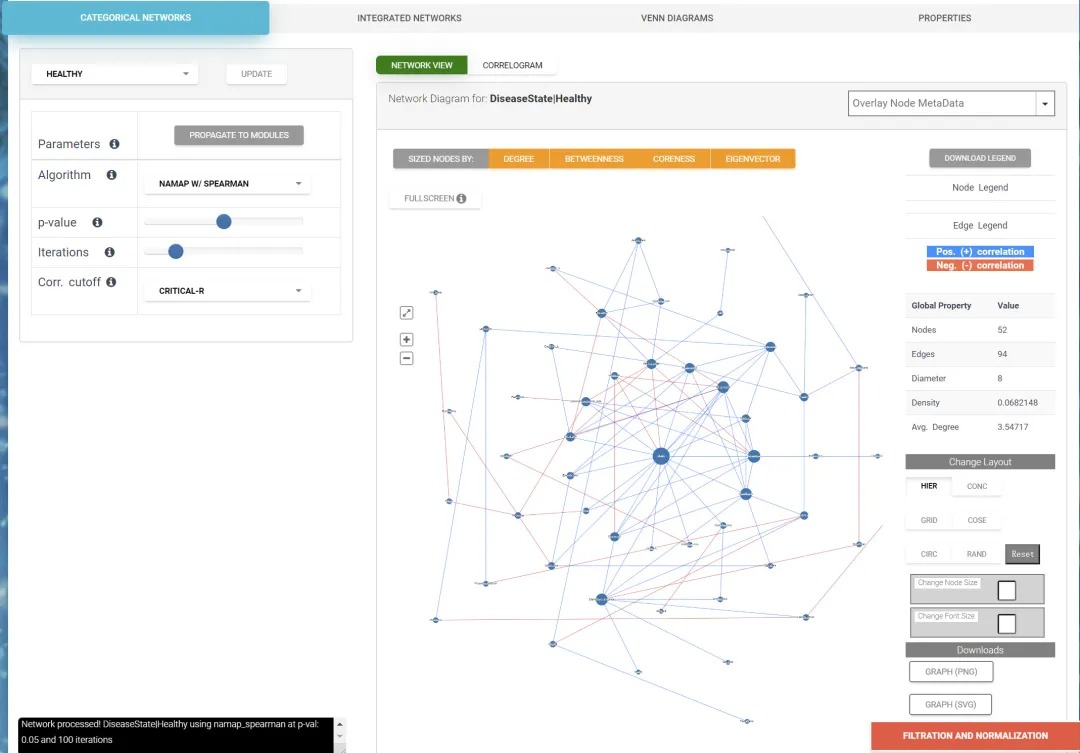
左侧为分组,参数选择面板;
右侧为图像预览区;
点大小,可按度、介数、紧密性、特征向量(eigenvector)显示;
点可按分类学着色,
边按正、负着色;
还有网络属性值,结点、边、半径、密度、平均度;
布局(Layout): 有6种方案可选,包括 HIER(层级,Hierachical,有点凌乱 )、CONC(同心圆,Concentric,三圈圆形分布,按结点大小由外到内排序,推荐)、GRID(网格,按结点大小排序,适合矩阵区域展示)、COSE(中心紧密型)、CIRC(圆形,规则,但边太长,浪费中心空间)、RAND(随机,Random,明显重叠);
下载:有位图PNG、矢量图SVG、边列表Edgelist(边边和正负相关的在三列表)、Gephi的GDF文件(所有边点信息,包括末相关的点)、Cytoscape的Jason文件(包括点id、名称、颜色、大小;边的源-目标和颜色)、以及丰度表(Abundance table,即按组筛选后、标准化的特征表)
整合网络(Integrated networks)
整合网络可以添加特征表中额外的数据,如元数据中的连续型变量、以及第二个特征表(如功能组成)。而且每种数据可多选。
这里我们选择元数据中的年龄(age),额外表中选择糖酵解(Glycolysis_Gluconeogenesis)和三羧酸循环(TCA),点击update,布局切换为自己喜欢的样式,如CONC或GRID,点击下载SVG,在新窗口中可再次调整布局,增大点和文字大小到合适位置,观察相关关系。
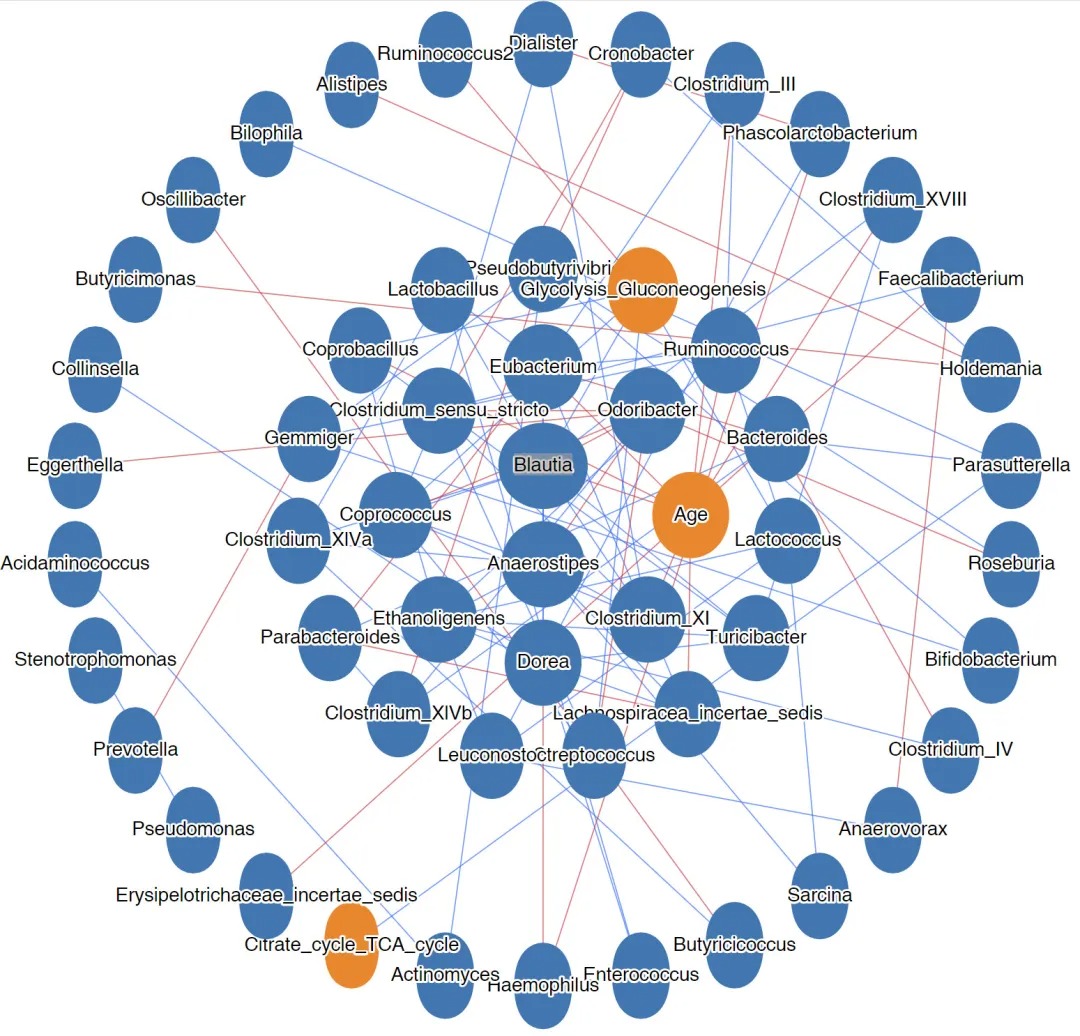
注:网站有bug,经常出现文字大小无法修改大小。可以下载矢量图SVG,在新窗口中可以调整,或下载后使用如Adobe Illustrator等矢量图编辑工具调整。
维恩图(Venn diagrams)
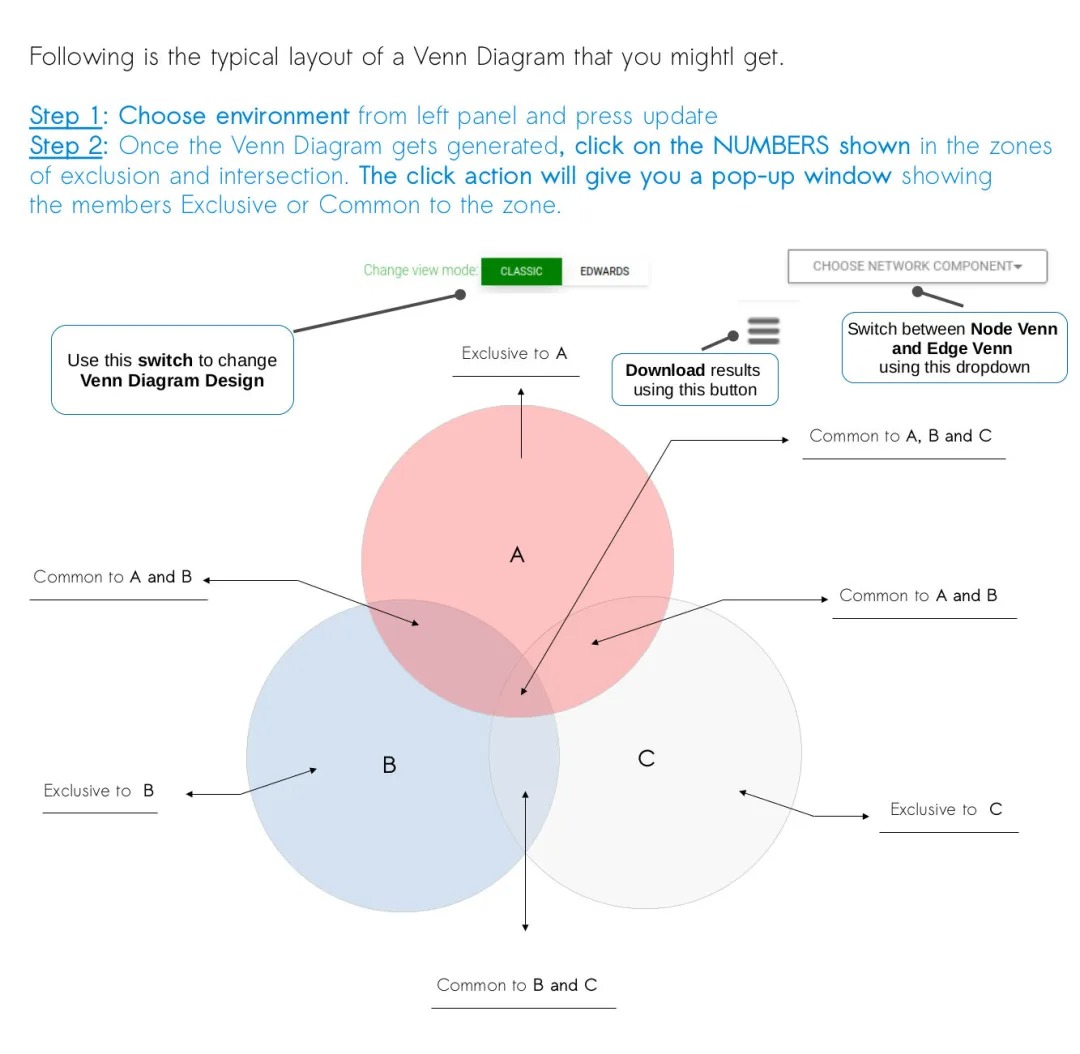
选择分组类型,如选择DiseaseState,再点击Update。出现4个组的维恩比较。这4组将不能进一步选择,如需要调整可能需要在上传前筛选元数据。
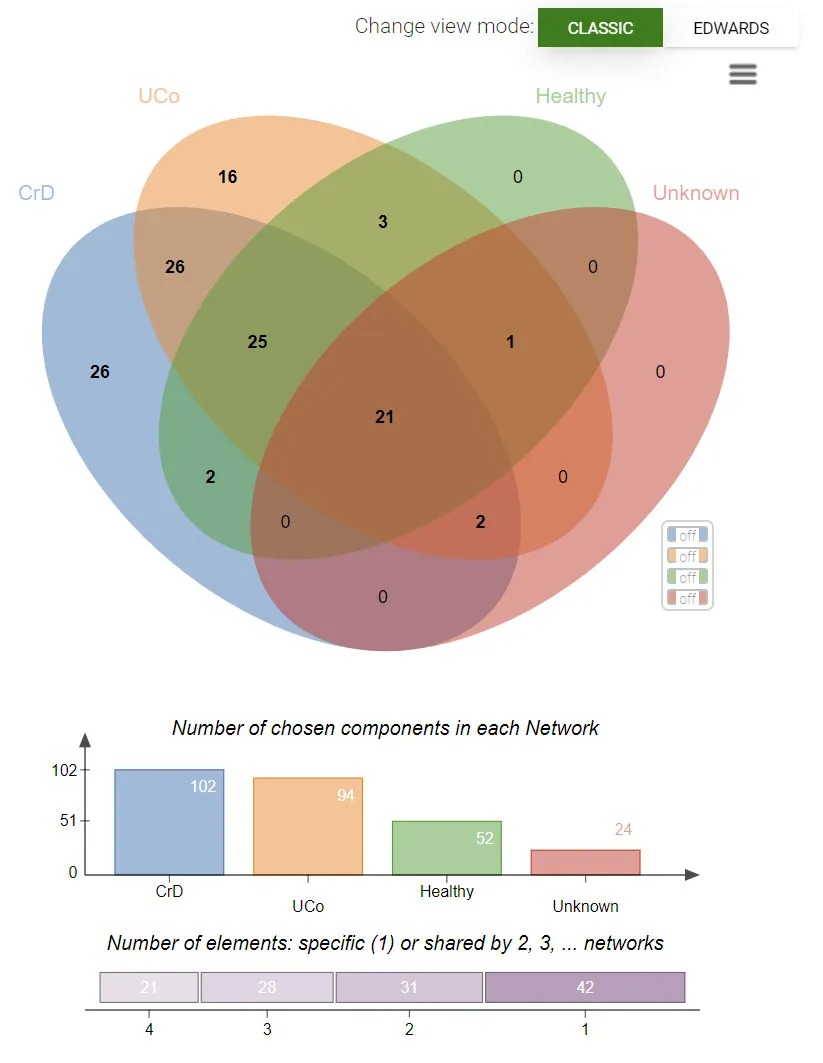
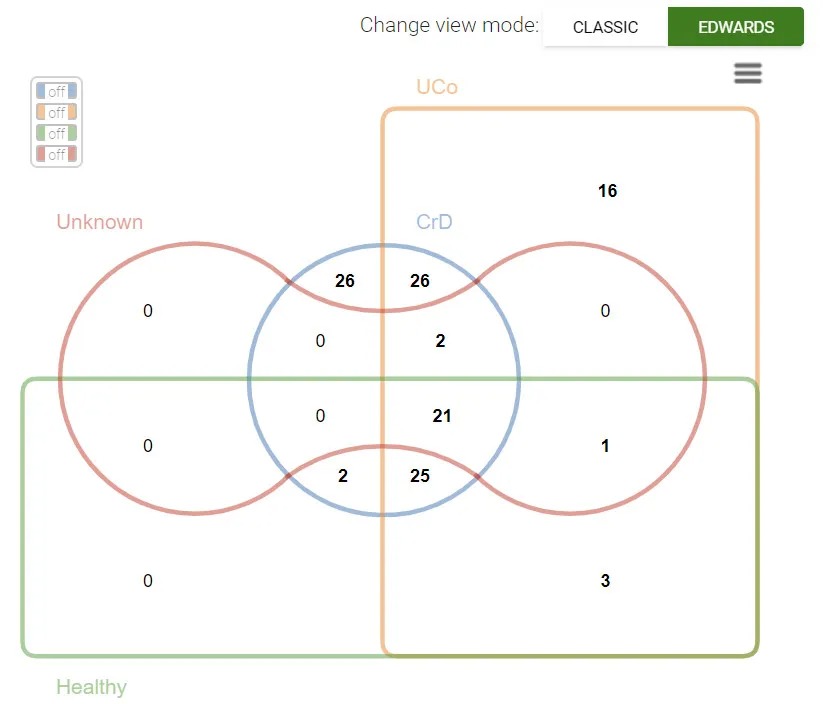
图中包括韦恩图、各种在网络中的总结点数量柱状图、不同组共享的结点数量热图。默认为经典(Classic)模式,可以切换为EDWARDS模式,如下图所示:
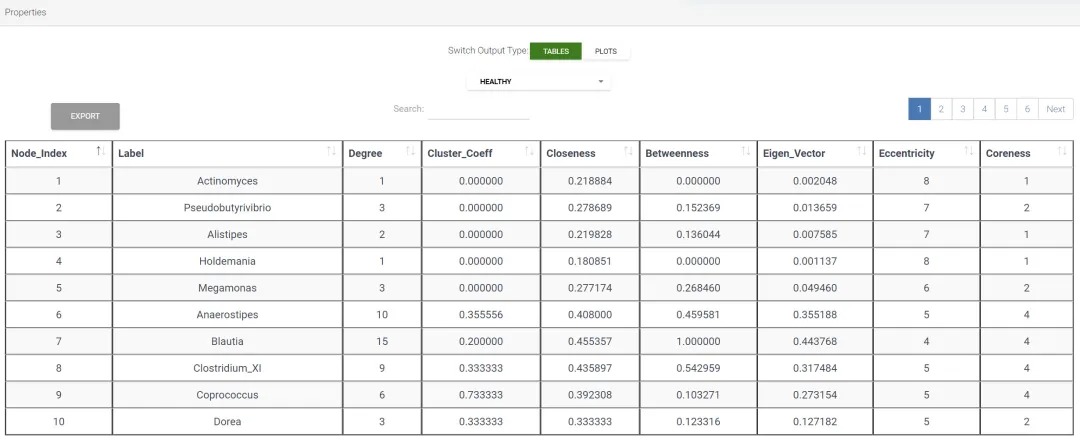
网络属性(Properties)
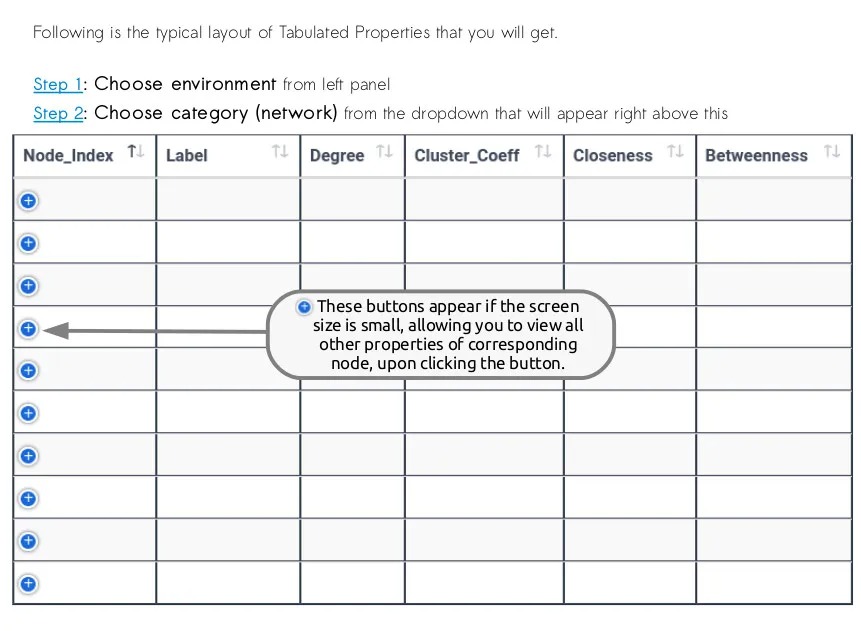
选择分组类
再选择分组中的一组,即显示网络属性
表可以进一步搜索(Search)和导出(Export)
点击PLOTS切换至绘图:
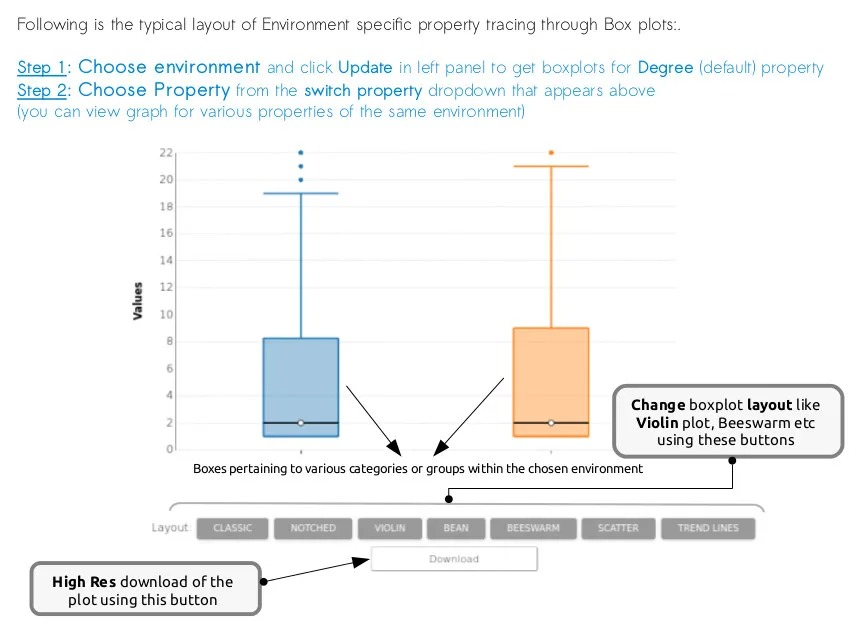
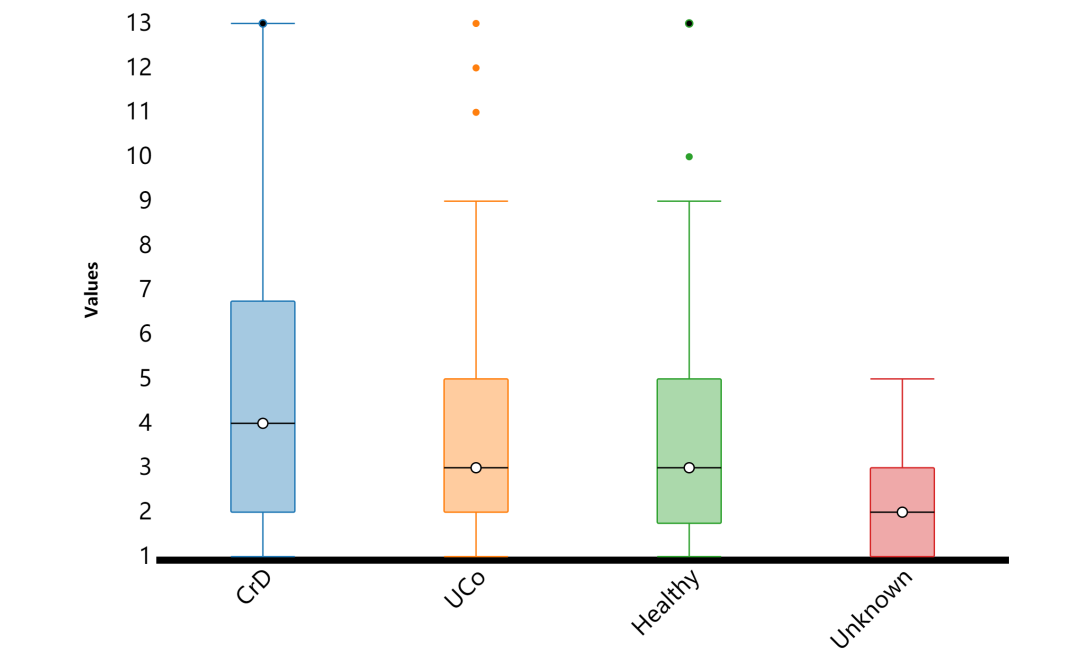
点击Update更新,展示度的组间箱线图比较;
该图形可切换不同属性,也有7种样式选择,但只能下载png位图;
数据实战
准备输入文件
属丰度表,一般需要筛选,不然结点太多看不清。比如我的属水平有500多个条目,我用R语言筛选均值大于千分之一的属有70个属(也可以在Excel中求均值再排序),也可以在线分析时灵活筛选;
样本元数据,包括样本的分组和其他数值型属性;
属水平高级分类,一般为属对应的门、纲、目、科四级信息。注:可以考虑把未分类属去掉,不然层级注释会有1对多的问题。
网站提交
访问主页:https://web.rniapps.net/metagenonets/
点击:GOTO: SUBMISSION SECTION
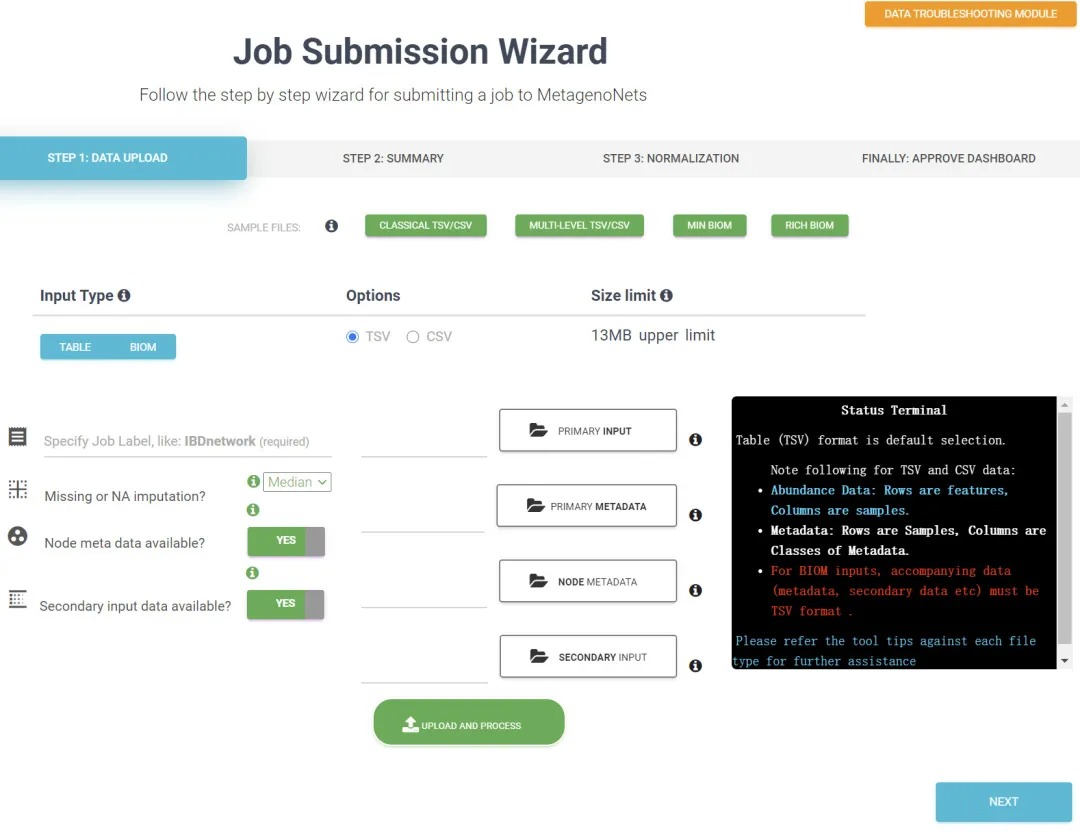
填写任务名称(Specify Job Label):随便写,如Project1
属级别特征表(PRIMARY INPUT)
样本元数据(PRIMARY METADATA)
属水平高级分类(NODE METADATA)
其他特征表(SECONDARY INPUT),没有可不填
点击上传和处理“UPLOAD AND PROCESS”,再点右下角下一步“NEXT”
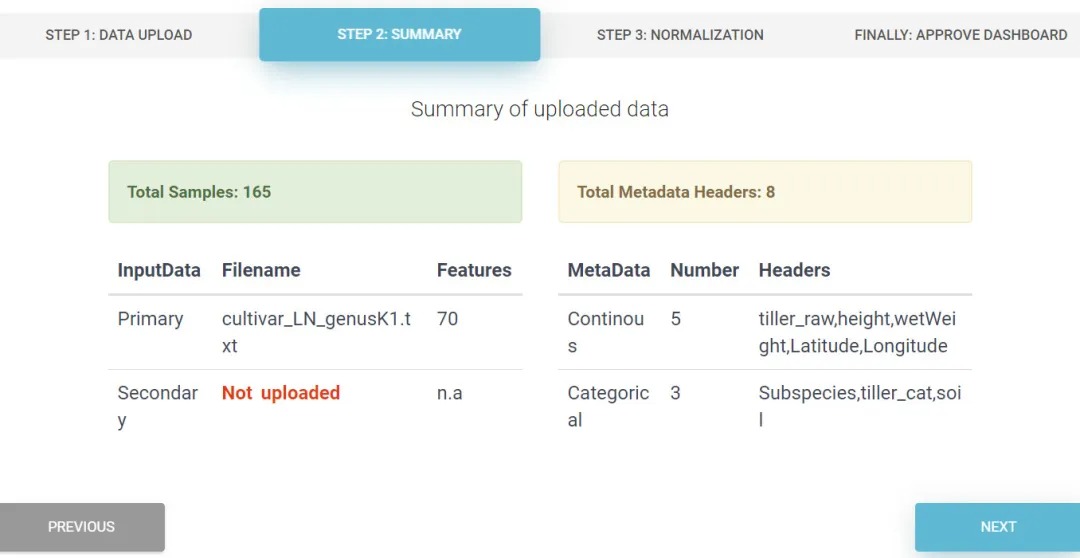
会显示数据概述信息,核对信息是否识别正确,再点右下角下一步“NEXT”。
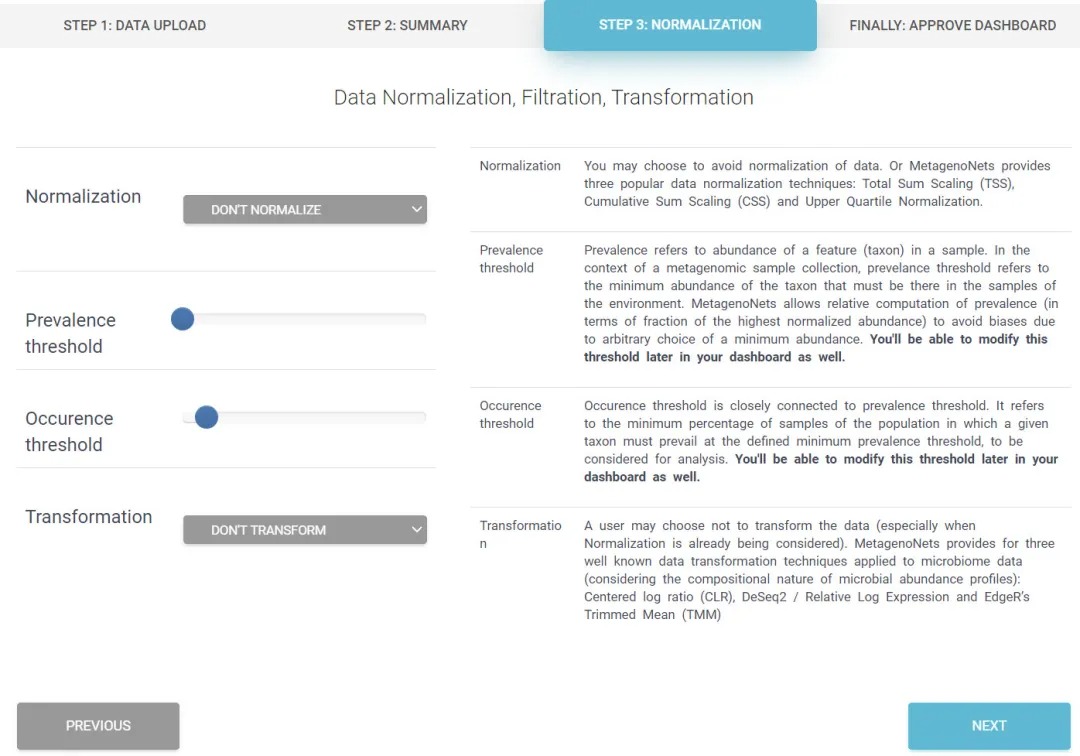
数据标准化。这里默认就好。具体可按自己的数据类型选择标准化方法如下:
标准化(Normalization):可选总体、CSS和分位数标准化;这里我们数据我们已经做过总体标准化,因此采用默认不标准化(DON”T NORMALIZE)
流行度阈值(Prevalance threshold):即丰度阈值,默认为0.0001,即万分之一,此值对网络图中结果数量控制非常有效;
出现频率(Occurence threshold):默认在至少10%的样本中出现;可有效过滤低频特征;
数据转换(Transformation):支持CLR、TMM等转换。对结果相关性很差时可尝试转换重试,这里使用默认不转换。
再点右下角下一步“NEXT”。
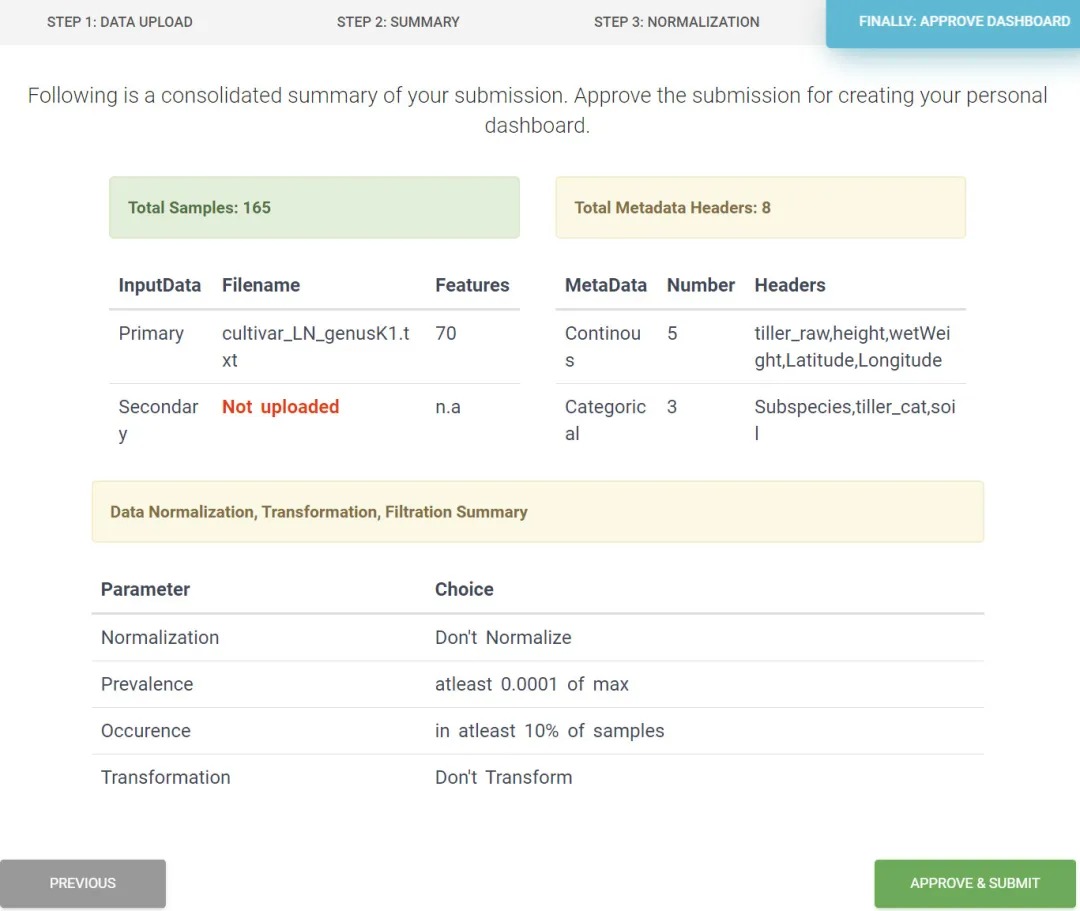
以上信息再次确定 ,再点右下角下一步“APPROVE & SUBMIT”。开始分析,接下来分析步骤同上面示例数据演示。
分析中的一些经验和问题
算法(Algorithm):默认为Spearman相关分析,我的结果是有正,有负相关;而且SPARCC分析时,结果相关全为正,不符合之前分析的预期。一般方法是没有优劣之分,而是要根据背景知识来判断结果是否合理,不同类型的数据可能需要不同的方法才有科学合理的结果。
p-value过滤: 输入70个属,Spearman相关方法下,默认P值0.05时,68个属,683条边,边多太看不清关系;修改为0.01时,点Update更新,54个点,129个边,阅读性较强;如果还是太多看不清,可进一步提交阈值。
常见问题
切换布局后,修改字体(Font Size)有时会不可用;可选择下载SVG,在弹出新窗口中可以选择布局和调整字体大小。
网络属性计算一次后,再修改参数结果也不再更新,有时需要重新建立任务重新计算。














